Haplogroupe N (Y-ADN)
En génétique humaine, l’haplogroupe N (LLY22G) est un haplogroupe du chromosome Y, présent surtout dans le nord de l'Eurasie. Il serait apparu il y a environ 20 000 ans en Asie du Sud-Est, avant de se répandre plus tard en Asie du Nord-Est, puis de gagner l'Europe du Nord à travers la Sibérie au cours du IIIe millénaire av. J.-C., en véhiculant les langues ouraliennes.

Origine
modifierOn estime que l'haplogroupe NO-M214 (le dernier ancêtre commun de l'haplogroupe N et de l'haplogroupe O-M175) est apparu entre 36 800 et 44 700 ans avant le présent (AP)[2],[3],[4].
L'haplogroupe N-M231 serait apparu en Asie du Sud-Est il y a environ 19 400 ans (± 4 800 ans), puis il se serait répandu vers le nord de l'Asie orientale au cours du Tardiglaciaire. Des hommes portant ce génome ont apparemment migré vers le nord alors que le climat se réchauffait progressivement, traversant la Chine et s'implantant en Mandchourie. L'haplogroupe N était prépondérant dans le Nord-Est de la Chine durant le Néolithique, il y a environ 6 500 ans, puis a décru graduellement dans cette région jusqu'à l'Âge du bronze, il y a environ 2 700 ans.
La rareté de l'haplogroupe N-M231 chez les Amérindiens confirme son arrivée relativement tardive en Asie du Nord-Est[5].
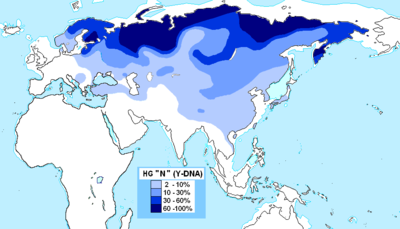
Diffusion vers l'Ouest
modifierAu cours du IIIe millénaire av. J.-C., l'haplogroupe N se diffuse d'Est en Ouest à travers la Sibérie, jusqu'à atteindre des régions aussi éloignées que la Fennoscandie et les Pays baltes[6].
Les résultats des études archéogénétiques suggèrent que la diffusion des langues ouraliennes vers l'Europe du Nord-Est vers s'est produite à partir de la population de Iakoutie de la fin du Néolithique. La population iakoute de cette période possède en effet tous les sous-clades de l'haplogroupe N, que l'on trouve aussi dans les populations ouraliennes[7]. Tous les hommes étudiés (en 2023) de la population iakoute de cette période appartiennent à l'haplogroupe N. Celle-ci est génétiquement liée à toutes les populations de langues ouraliennes anciennes et contemporaines[7].
La diffusion de l'haplogroupe N vers l'Ouest à partir de coïnciderait avec un réchauffement climatique dans le nord de l'Eurasie. Elle parait liée à l'expansion des locuteurs de langues ouraliennes[8].
Sous-groupes
modifierLes sous-clades N3 et N2a de l'haplogroupe N sont les plus fréquents. Ils ont divergé il y a environ 18 000 ans[8].
L'une des caractéristiques génétiques les plus remarquables des populations de la Baltique orientale est un haplogroupe N3a à haute fréquence, une caractéristique partagée principalement avec les groupes de langue finno-ougrienne en Europe et plusieurs populations dans toute la Sibérie. L’expansion rapide de ce sous-clade a probablement eu lieu au cours des 5 000 dernières années[9]. Les locuteurs ouraliens seraient arrivés dans la Baltique orientale seulement au Ier millénaire av. J.-C. Plus d'un tiers des hommes estoniens modernes partagent l’haplogroupe N3a, commun dans d'autres populations de langue ouralienne vivant beaucoup plus à l'est, mais qui est introuvable aux époques antérieures à l'Âge du fer dans la Baltique orientale[9].
Miroslava Derenko et collègues ont noté qu'il existe deux sous-groupes au sein du sous-clade N3a, tous deux présents en Sibérie et en Europe du Nord, avec des histoires différentes. Celui qu'ils ont appelé N3a1 s'est d'abord développé en Sibérie méridionale puis s'est étendu vers le Nord de l'Europe. Pendant ce temps, le sous-groupe qu'ils ont appelé N3a2 a émergé dans le Sud de la Sibérie (probablement dans la région du lac Baïkal)[10].
Le sous-groupe N3a1a-M178 est défini par la présence des marqueurs M178 et P298. La fréquence moyenne du N-M178* est plus élevée en Europe du Nord qu'en Sibérie, atteignant environ 60 % chez les Finlandais et environ 40 % chez les Lettons, les Lituaniens et 35 % chez les Estoniens[10],[11].
En Europe, le sous-clade N3a3-VL29 représente environ un tiers des Estoniens, Lettons et Lituaniens. Il est également présent chez les Samis, les populations de Carélie et les Finnois.
Références
modifier- (en) H Shi, X Qi, H Zhong, Y Peng, X Zhang et al., « Genetic Evidence of an East Asian Origin and Paleolithic Northward Migration of Y-chromosome Haplogroup N », PLOS One, vol. 8, no 6, , e66102 (PMID 23840409, PMCID 3688714, DOI 10.1371/journal.pone.0066102, Bibcode 2013PLoSO...866102S, lire en ligne)
- « arbre génétique des haplogroupes du chromosome Y », sur YFull, v6.05.11 au 25 septembre 2018
- (en) Monika Karmin, Lauri Saag, Mário Vicente et al., « A recent bottleneck of Y chromosome diversity coincides with a global change in culture », Genome Research, vol. 25, no 4, , p. 459–466 (DOI 10.1101/gr.186684.114)
- (en) G. David Poznik, Yali Xue, Fernando L. Mendez et al., « Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences », Nature Genetics, vol. 48, no 6, , p. 593–599 (DOI 10.1038/ng.3559)
- Chiaroni, Underhill et Cavalli-Sforza 2009.
- Rootsi 2006.
- (en) Tian Chen Zeng, Leonid A. Vyazov, Alexander Kim et al., Postglacial genomes from foragers across Northern Eurasia reveal prehistoric mobility associated with the spread of the Uralic and Yeniseian languages, biorxiv.org, 4 octobre 2023, doi.org/10.1101/2023.10.01.560332
- (en) Anne-Mai Ilumäe et al., Human Y Chromosome Haplogroup N: A Non-trivial Time-Resolved Phylogeography that Cuts across Language Families, AJHG, volume 99, numéro 1, p.163-173, 7 juillet 2016
- (en) Lehti Saag, Margot Laneman, Liivi Varul et Martin Malve, « The Arrival of Siberian Ancestry Connecting the Eastern Baltic to Uralic Speakers further East », Current Biology, vol. 29, no 10, , p. 1701–1711.e16 (ISSN 0960-9822, DOI 10.1016/j.cub.2019.04.026, lire en ligne)
- Derenko 2007.
- Lappalainen 2008.
Bibliographie
modifier- (en) Siiri Rootsi et al., « A counter-clockwise northern route of the Y-chromosome haplogroup N from Southeast Asia towards Europe », European Journal of Human Genetics, vol. 15, no 2, , p. 204–211 (PMID 17149388, DOI 10.1038/sj.ejhg.5201748)
- (en) Miroslava Derenko, Boris Malyarchuk, Galina Denisova, Marcin Wozniak, Tomasz Grzybowski, Irina Dambueva et Ilia Zakharov, « Y-chromosome haplogroup N dispersals from south Siberia to Europe », Journal of Human Genetics, vol. 52, no 9, , p. 763–770 (PMID 17703276, DOI 10.1007/s10038-007-0179-5)
- (en) T. Lappalainen, V. Laitinen, E. Salmela, P. Andersen, K. Huoponen, M.-L. Savontaus et P. Lahermo, « Migration Waves to the Baltic Sea Region », Annals of Human Genetics, vol. 72, no 3, , p. 337–348 (PMID 18294359, DOI 10.1111/j.1469-1809.2007.00429.x)
- (en) Jacques Chiaroni, Peter A. Underhill et Luca L. Cavalli-Sforza, « Y chromosome diversity, human expansion, drift, and cultural evolution », Proceedings of the National Academy of Sciences, vol. 106, no 48, , p. 20174–20179 (PMID 19920170, PMCID 2787129, DOI 10.1073/pnas.0910803106, Bibcode 2009PNAS..10620174C)
Articles connexes
modifier|
Haplogroupes du chromosome Y (Y-ADN) | ||||||||||||||||||||||||
| Plus récent ancêtre patrilinéaire commun | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| G | H | IJK | ||||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| I1 | L | T | MS | P | NO | |||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||