Paléopolyploïdie
La paléopolyploïdie fait référence à une polyploïdie ancestrale survenue il y a plusieurs millions d'années à la suite d'une duplication complète du génome par autopolyploïdie ou bien allopolyploïdie. Cette duplication du génome donne un organisme avec 4n lots de chromosomes ou plus. Cet état est transitoire car en raison de la redondance fonctionnelle des gènes, les gènes (ohnologues) sont rapidement éteints ou massivement éliminés par le phénomène de fractionnement des génomes. La plupart des génomes paléoploïdes au cours de l'évolution ont perdu cette polyploïdie apparente par la perte des gènes redondants. Ce processus s'appelle la diploïdisation. De plus, les gènes ohnologues divergent en accumulant des mutations qui aboutissent à une subfonctionnalisation ou bien à une néofonctionnalisation. La recherche actuelle s'efforce de déterminer si cette perte de gènes est aléatoire ou biaisée[4].
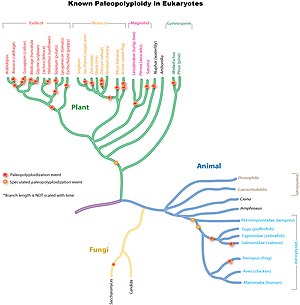
Par exemple, de nos jours, la levure du boulanger Saccharomyces cerevisiae, Arabidopsis thaliana et les poissons téléostéens sont des organismes diploïdes alors qu'au cours de leur évolution ils ont subi une ou plusieurs duplications complètes du génome. Ils sont donc paléopolyploïdes.
Voir aussi
modifierRéférences
modifier- (en) Kenneth H. Wolfe, « Yesterday's polyploids and the mystery of diploidization », Nature Reviews Genetics, vol. 2, no 5, , p. 333–341 (ISSN 1471-0056, DOI 10.1038/35072009, résumé).
- (en) Keith L. Adams et Jonathan F. Wendel, « Polyploidy and genome evolution in plants », Current Opinion in Plant Biology, vol. 8, no 2, , p. 135–141 (ISSN 1369-5266, DOI 10.1016/j.pbi.2005.01.001, résumé).
- (en) Liying Cui, P. Kerr Wall, James H. Leebens-Mack, Bruce G. Lindsay, Douglas E. Soltis, Jeff J. Doyle, Pamela S. Soltis, John E. Carlson, Kathiravetpilla Arumuganathan, Abdelali Barakat, Victor A. Albert, Hong Ma et Claude W. dePamphilis, « Widespread genome duplications throughout the history of flowering plants », Genome Research, vol. 16, no 6, , p. 738–749 (ISSN 1088-9051, DOI 10.1101/gr.4825606, lire en ligne).
- (en) Richard J Langham, Justine Walsh, Molly Dunn, Cynthia Ko, Stephen A Goff & Michael Freeling, « Genomic duplication, fractionation and the origin of regulatory novelty », Genetics, vol. 166, no 2, , p. 935-945.